- 448235-52-7
-
Pyrrolysine
Pyrrolysine 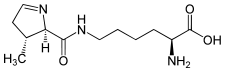
Structure de la pyrrolysineGénéral Nom IUPAC N6-[(2R,3R)-3-méthyl-3,4-dihydro-2H-pyrrol-2-ylcarbonyl]-L-lysine Synonymes O (Pyl) No CAS PubChem SMILES Propriétés chimiques Formule brute C12H21N3O3 Masse molaire 255,3134 g∙mol-1
C 56,45 %, H 8,29 %, N 16,46 %, O 18,8 %,Propriétés biochimiques Codons Codon-STOP « ambre » UAG avec séquence d'insertion PylIS Unités du SI & CNTP, sauf indication contraire. La pyrrolysine est un acide aminé naturel possédant une correspondance dans le code génétique utilisé par certaines archées[1] méthanogènes. Il est principalement rencontré dans les enzymes faisant partie du métabolisme responsable de la production de méthane. Son nom complet en nomenclature IUPAC est N6-[(2R,3R)-3-methyl-3,4-dihydro-2H-pyrrol-2-ylcarbonyl]-L-lysine. On recommande de désigner cet acide aminé par le trigramme Pyl et le symbole à une lettre O.
Sommaire
Expression génétique de la pyrrolysine
Ce dérivé de la lysine est codé par le codon UAG (codon-STOP « ambre » chez la majorité des autres organismes). Ce codon est probablement modifié par la présence d'une séquence spécifique en aval, nommée PYLIS[2], qui forme une structure en épingle à cheveux (ou « tige-boucle ») dans l'ARNm, forçant l'incorporation d'un résidu pyrrolysine au lieu de terminer la traduction.
Il est également intéressant de noter que le codon-STOP UAG semble être utilisé beaucoup moins souvent que les autres codons-STOP et que, lorsque il est rencontré dans une structure ouverte en lecture, il est toujours suivi peu après par un ou plusieurs des deux autres codon-STOP.
Proche d'un groupe (cluster) de gènes de la méthyltransférase de Methanosarcina barkeri se trouve le gène pylT, qui code un ARN de transfert (ARNt) inhabituel avec un anticodon CUA. Le gène pylS adjacent encode une aminoacyl-ARNt synthétase de classe II qui charge l'ARNt dérivé du pylT avec la pyrrolysine.
L'opéron contenant pylT et pylS se trouve également dans le génome séquencé d'autres membres de la famille Methanosarcinaceae. Des homologues de pylS and pylT ont été décrits dans la bactérie Gram-positive Desulfitobacterium hafniense. La fonction de ces gènes putatifs dans ces organismes demeure inconnue. Il a été démontré à l'origine que l'ARNt (CUA) codé par pylT peut se charger de la lysine par PylS. Plus récemment, il a été montré que l'ARNt (CUA) peut être chargé avec la lysine in vitro avec l'action concertée des Lysyl-ARNt synthétases de classe I et de classe II de M. barkeri. Le chargement d'un ARNt (CUA) avec la lysine est l'hypothèse originelle de la première étape dans la traduction des codons UAG (ambre) en pyrrolysine chez certains méthanogènes. Le modèle actuel fondé sur des données in vitro et in vivo est en faveur de l'hypothèse du chargement direct de la pyrrolysine sur l'ARNt (CUA) par la protéine produite par le gène pylS. Ceci fait de la pyrrolysine le 22ème acide aminé naturel codé génétiquement.
Notes et références
Voir aussi
- (en) Cet article est partiellement ou en totalité issu d’une traduction de l’article de Wikipédia en anglais intitulé « Pyrrolysine ».
Articles connexes
- Code génétique
- Traduction génétique
- Sélénocystéine, le 21ème acide aminé codé génétiquement.
Liens externes
Bibliographie
- Portail de la chimie
- Portail de la biochimie
- Portail de la biologie cellulaire et moléculaire
Catégories : Acide aminé | Métabolisme de l'azote | Azoline
Wikimedia Foundation. 2010.
